| treat | replicate 1 | replicate 2 | replicate 3 |
|---|---|---|---|
| Fertilizer | 0.020 | -0.217 | -0.273 |
| F. open | 0.940 | 0.780 | 0.555 |
| F.+sugar | 0.188 | -0.100 | 0.020 |
| F.+CaCO3 | 0.245 | 0.236 | 0.456 |
| Bas.med. | 0.699 | 0.727 | 0.656 |
| A.dest | -0.010 | 0.000 | -0.010 |
| Tap water | 0.030 | -0.070 | NA |
07-ANOVA und ANCOVA
Angewandte Statistik – Ein Praxiskurs
2025-12-13
ANOVA - Varianzanalyse
- Prüfung komplexer Hypothesen als Ganzes, z.B.:
- mehr als zwei Stichproben (Mehrfachtestproblem),
- mehrere multiple Faktoren (Mehrfach- ANOVA)
- Eliminierung von Kovariaten (ANCOVA)
- feste und/oder zufällige Effekte (Varianzzerlegung, Modelle mit gemischten Effekten)
- Unterschiedliche Anwendungsszenarien:
- explorative Anwendung: Welche Einflussfaktoren sind wichtig?
- Signifikanztests
- statistische Modellierung: Quantifizierung von Effekten und Abhängigkeiten.
- ANOVA-Verfahren basieren (meistens) auf linearen Modellen.
Ein Praxisbeispiel
Suche nach einem geeigneten Medium für Wachstumsexperimente mit Grünalgen
- Schulprojekt aus “Jugend Forscht”
- geeignet für Kurse in Schule und Studium
- preisgünstig, einfach zu handhaben

Idee
- Verwendung eines kommerziellen Düngers mit den Hauptnährstoffen N und P
- Mineralwasser mit Spurenelementen
- Enthält stilles Mineralwasser genügend \(\mathrm{CO_2}\)?
- testen, wie man die Verfügbarkeit von \(\mathrm{CO_2}\) für die Photosynthese verbessern kann
Anwendung
7 Verschiedene Behandlungen
- Düngemittellösung in geschlossenen Flaschen
- Düngemittellösung in offenen Flaschen (\(\mathrm{CO_2}\) aus der Luft)
- Düngemittel + Zucker (organische C-Quelle)
- Dünger + zusätzliches \(\mathrm{HCO_3^-}\) (Zusatz von \(\mathrm{CaCO_3}\) zu sprudelndem Mineralwasser)
- Standard-Algenwachstumsmedium („Basalmedium“) zum Vergleich
- Deionisiertes („destilliertes“) Wasser und
- Leitungswasser zum Vergleich
Versuchsaufbau

- jede Behandlung mit 3 Wiederholungen
- randomisierte Platzierung auf dem Schüttler
- 16:8 Licht:Dunkel-Zyklus
- Transmissionsmessung direkt in den Flaschen mit einem selbstgebauten Messgerät
Ergebnisse
 Dünger – offene Flasche – + Zucker – + CaCO3 – Basalmedium – A. dest – Leitungswasser
Dünger – offene Flasche – + Zucker – + CaCO3 – Basalmedium – A. dest – Leitungswasser
Der Datensatz
- Wachstum von Tag 2 bis Tag 6 (relative Einheiten)
- NA bedeutet „nicht verfügbar“, d.h. ein fehlender Wert
- Die Kruztabelle ist kompakt und übersichtlich, jedoch nicht ideal für die Datenanalyse.
- \(\Rightarrow\) Konvertierung in das Langformat
Daten im Langformat
Vorteile
- sieht „unpraktisch“ aus, ist aber viel besser für die Datenanalyse
- abhängige Variable Wachstum und
Erklärungsvariable Behandlung deutlich sichtbar - Modellformel:
growth ~ treat - leicht erweiterbar auf \(>1\) Erklärungsvariable
| treat | rep | growth |
|---|---|---|
| Fertilizer | 1 | 0.020 |
| Fertilizer | 2 | -0.217 |
| Fertilizer | 3 | -0.273 |
| F. open | 1 | 0.940 |
| F. open | 2 | 0.780 |
| F. open | 3 | 0.555 |
| F.+sugar | 1 | 0.188 |
| F.+sugar | 2 | -0.100 |
| F.+sugar | 3 | 0.020 |
| F.+CaCO3 | 1 | 0.245 |
| F.+CaCO3 | 2 | 0.236 |
| F.+CaCO3 | 3 | 0.456 |
Die Daten in R
algae <- data.frame(
treat = factor(c("Fertilizer", "Fertilizer", "Fertilizer",
"F. open", "F. open", "F. open",
"F.+sugar", "F.+sugar", "F.+sugar",
"F.+CaCO3", "F.+CaCO3", "F.+CaCO3",
"Bas.med.", "Bas.med.", "Bas.med.",
"A.dest", "A.dest", "A.dest",
"Tap water", "Tap water"),
levels=c("Fertilizer", "F. open", "F.+sugar",
"F.+CaCO3", "Bas.med.", "A.dest", "Tap water")),
rep = c(1, 2, 3, 1, 2, 3, 1, 2, 3, 1, 2, 3, 1, 2, 3, 1, 2, 3, 1, 2),
growth = c(0.02, -0.217, -0.273, 0.94, 0.78, 0.555, 0.188, -0.1, 0.02,
0.245, 0.236, 0.456, 0.699, 0.727, 0.656, -0.01, 0, -0.01, 0.03, -0.07)
)- Kleine Datensätze können direkt in den Code eingebunden werden.
- Eine csv-Datei im Langformat ist ebenfalls möglich.
- Wichtig:
treatals Faktor (nominale Variable). - Mit
levelskann man die Reihenfolge fixieren.
Boxplot

Streifendiagramm

Besser als Boxplot, denn wir haben nur 2-3 Wiederholungen. Boxplot braucht mehr.
Umwandlung einer wissenschaftlichen Frage in eine statistische Hypothese
Wissenschaftliche Fragen
- Sind die Behandlungen unterschiedlich?
- Welches Medium ist das beste?
- Ist das beste Medium signifikant besser als die anderen?
- Wieviel besser ist das beste Medium (Efektstärke)?
Statistische Hypothesen
- \(H_0\): das Wachstum ist bei allen Behandlungen gleich
- \(H_A\): Unterschiede zwischen den Medien
Warum nicht einfach mehrere t-Tests?
- Wenn wir bei 7 Behandlungen jede gegen jede testen wollen, brauchen wir:
\[7 \cdot (7 - 1) / 2 = 21 \qquad\text{Tests.}\]
- Bei \(\alpha = 0.05\), erhält man 5% falsch positive Ergebnisse.
\(\Rightarrow\) Einer von 20 Tests ist im Durchschnitt falsch positiv. - Bei \(N\) Tests erhöht sich der Gesamtfehler von \(\alpha\) im schlimmsten Fall auf \(N\cdot\alpha\).
- Dies wird alpha-Fehler-Inflation oder Bonferroni-Ungleichung genannt:
\[ \alpha_{total} \le \sum_{i=1}^{N} \alpha_i = N \cdot \alpha \]
Wird die Regel ignoriert, erhält man zufällige falsch-positive Ergebnisse (statistical fishing).
Lösungsansätze
- Korrektur der Alpha-Fehler, so dass \(\alpha_{total} = 0.05\) bleibt;
\(\rightarrow\) Bonferroni-Regel. - Verfahren, das das Gesamtproblem behandelt:
\(\rightarrow\) ANOVA.
ANOVA: Varianzanalyse
Grundgedanke
- Aufteilung der Gesamtvarianz in Wirkung(en) und Fehler:
\[ s_y^2 = s^2_\mathrm{effect} + s^2_{\varepsilon} \]
- Etwas überraschend: Analyse von Varianzen, um Mittelwerte zu vergleichen.
- Erklärung: Mittelwertunterschiede tragen zur Gesamtvarianz der ganzen Stichprobe bei.
- Die Varianzkomponenten können als Varianz innerhalb (\(s^2_\varepsilon\)) und Varianz zwischen Stichproben bezeichnet werden.
- Die Varianz-Zerlegung erfolgt über ein lineares Modell.
Beispiel
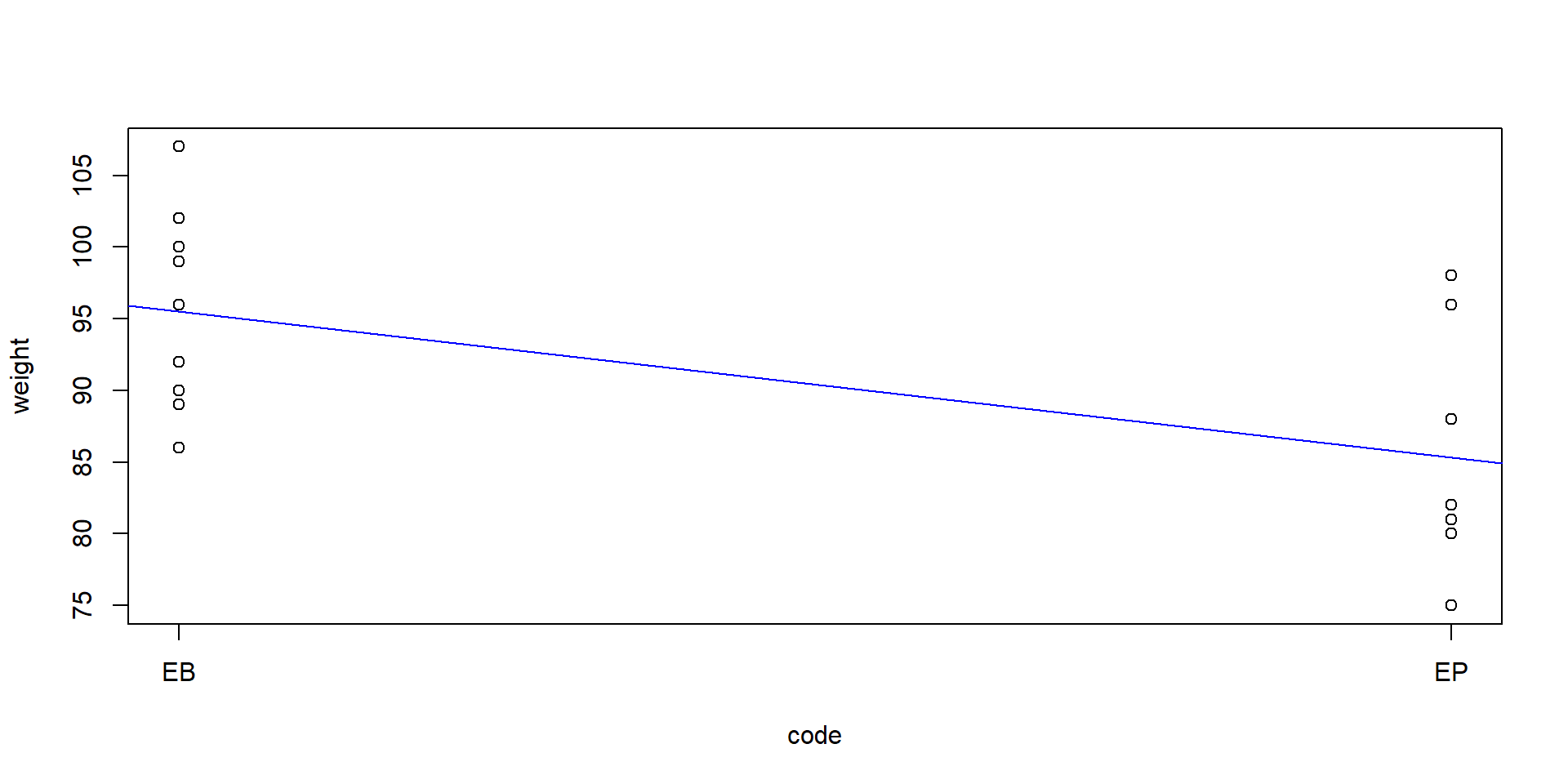
Zwei Marken von Clementinen aus einem Geschäft „E“, die wir als „EB“ und „EP“ bezeichnen. Wir wollen wissen, ob die Premiummarke („P“) und die Basismarke („B“) ein unterschiedliches Gewicht haben.
Wir kodieren eine Stichprobe („EB“) mit 1 und die andere Stichprobe („EP“) mit 2:
Dann wird eine lineare Regression angewandt
Lineares Modell und Varianzkomponenten
Gesamtvarianz
Restvarianz (= Varianz innerhalb der Behandlungen)
Erklärte Varianz (= Varianz zwischen den Behandlungen)
- Das Beispiel zeigt das Grundprinzip.
- Aber: Die „erklärte Varianz“ ist nicht für den F-Test der ANOVA geeignet, weil die Freiheitsgrade nicht berücksichtigt sind.
Korrekte Berechnung mit Freiheitsgraden
# Freiheitsgrade
n <- nrow(clem)
k <- length(unique(clem$brand))
df_total <- n - 1 # -1 df für den Gesamtmittelwert (Intercept)
df_between <- k - 1 # df für Gruppenunterschiede (nach Berücksichtigung des Gesamtmittelwerts)
df_residuals <- n - k # -1 df pro Gruppe (insgesamt k Gruppen)
# Prüfung: df_total sollte gleich df_between + df_residuals sein
c(n = n, k = k, df_total = df_total, df_between = df_between, df_residuals = df_residuals) n k df_total df_between df_residuals
23 2 22 1 21 # Sums of squares
SS_total <- var_tot * df_total
SS_residuals <- var_res * df_total # var(residuals) nutzt n−1
SS_between <- SS_total - SS_residuals
# Mean sums of squares
MS_between <- SS_between / df_between
MS_residuals <- SS_residuals / df_residuals
c(MS_between = MS_between, MS_residuals = MS_residuals) MS_between MS_residuals
566.23913 45.30952 # F-Test
F_stat <- MS_between / MS_residuals
p_val <- pf(F_stat, df1 = df_between, df2 = df_residuals, lower.tail = FALSE)
c(F = round(F_stat, 3), p = round(p_val, 4)) F p
12.497 0.002 Die ANOVA arbeitet direkt mit den Summen der Quadrate, ohne den Umweg über die Varianzen.
ANOVA in einem Schritt
Analysis of Variance Table
Response: weight
Df Sum Sq Mean Sq F value Pr(>F)
code 1 566.24 566.24 12.497 0.001963 **
Residuals 21 951.50 45.31
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Ein t-Test zum Vergleich
Two Sample t-test
data: weight by code
t = 3.5351, df = 21, p-value = 0.001963
alternative hypothesis: true difference in means between group 1 and group 2 is not equal to 0
95 percent confidence interval:
4.185911 16.147423
sample estimates:
mean in group 1 mean in group 2
95.50000 85.33333 \(\Rightarrow\) Die p-Werte sind identisch.
- Bei zwei Gruppen ist die ANOVA (F-Test) äquivalent zum t-Test mit gleichen Varianzen.
- \(F = t^2\), \(p\) gleich.
ANOVA mit mehr als 2 Stichproben
Zurück zum Algenwachstums-Experiment:
- Wir könnten die Koeffizienten mit
summary(m)ausgeben. - Zunächst interessiert uns der Gesamteffekt, daher verwenden wir
anova(m).
Analysis of Variance Table
Response: growth
Df Sum Sq Mean Sq F value Pr(>F)
treat 6 2.35441 0.39240 25.045 1.987e-06 ***
Residuals 13 0.20368 0.01567
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1- zeigt den F-Test für den Gesamteffekt des Faktors
treat.
\(\Rightarrow\) Der p-Wert ist sehr klein → der Behandlungseffekt ist signifikant.
Posthoc-Tests
- Der F-Test zeigte, dass der Faktor „Behandlung“ einen signifikanten Effekt hat.
- Wir wissen jedoch noch nicht, welche spezifischen Gruppen sich unterscheiden.
- Tukey-HSD-Test (HSD = Honest Significant Difference): am häufigsten verwendeter Post-hoc-Test für ANOVA.
- vergleicht alle Paare von Gruppen und berücksichtigt die family-wise error rate.
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = m)
$treat
diff lwr upr p adj
F. open-Fertilizer 0.91500000 0.56202797 1.26797203 0.0000103
F.+sugar-Fertilizer 0.19266667 -0.16030537 0.54563870 0.5211198
F.+CaCO3-Fertilizer 0.46900000 0.11602797 0.82197203 0.0069447
Bas.med.-Fertilizer 0.85066667 0.49769463 1.20363870 0.0000231
A.dest-Fertilizer 0.15000000 -0.20297203 0.50297203 0.7579063
Tap water-Fertilizer 0.13666667 -0.25796806 0.53130140 0.8837597
F.+sugar-F. open -0.72233333 -1.07530537 -0.36936130 0.0001312
F.+CaCO3-F. open -0.44600000 -0.79897203 -0.09302797 0.0102557
Bas.med.-F. open -0.06433333 -0.41730537 0.28863870 0.9943994
A.dest-F. open -0.76500000 -1.11797203 -0.41202797 0.0000721
Tap water-F. open -0.77833333 -1.17296806 -0.38369860 0.0001913
F.+CaCO3-F.+sugar 0.27633333 -0.07663870 0.62930537 0.1727182
Bas.med.-F.+sugar 0.65800000 0.30502797 1.01097203 0.0003363
A.dest-F.+sugar -0.04266667 -0.39563870 0.31030537 0.9994197
Tap water-F.+sugar -0.05600000 -0.45063473 0.33863473 0.9985686
Bas.med.-F.+CaCO3 0.38166667 0.02869463 0.73463870 0.0307459
A.dest-F.+CaCO3 -0.31900000 -0.67197203 0.03397203 0.0879106
Tap water-F.+CaCO3 -0.33233333 -0.72696806 0.06230140 0.1247914
A.dest-Bas.med. -0.70066667 -1.05363870 -0.34769463 0.0001792
Tap water-Bas.med. -0.71400000 -1.10863473 -0.31936527 0.0004507
Tap water-A.dest -0.01333333 -0.40796806 0.38130140 0.9999997Grafische Darstellung
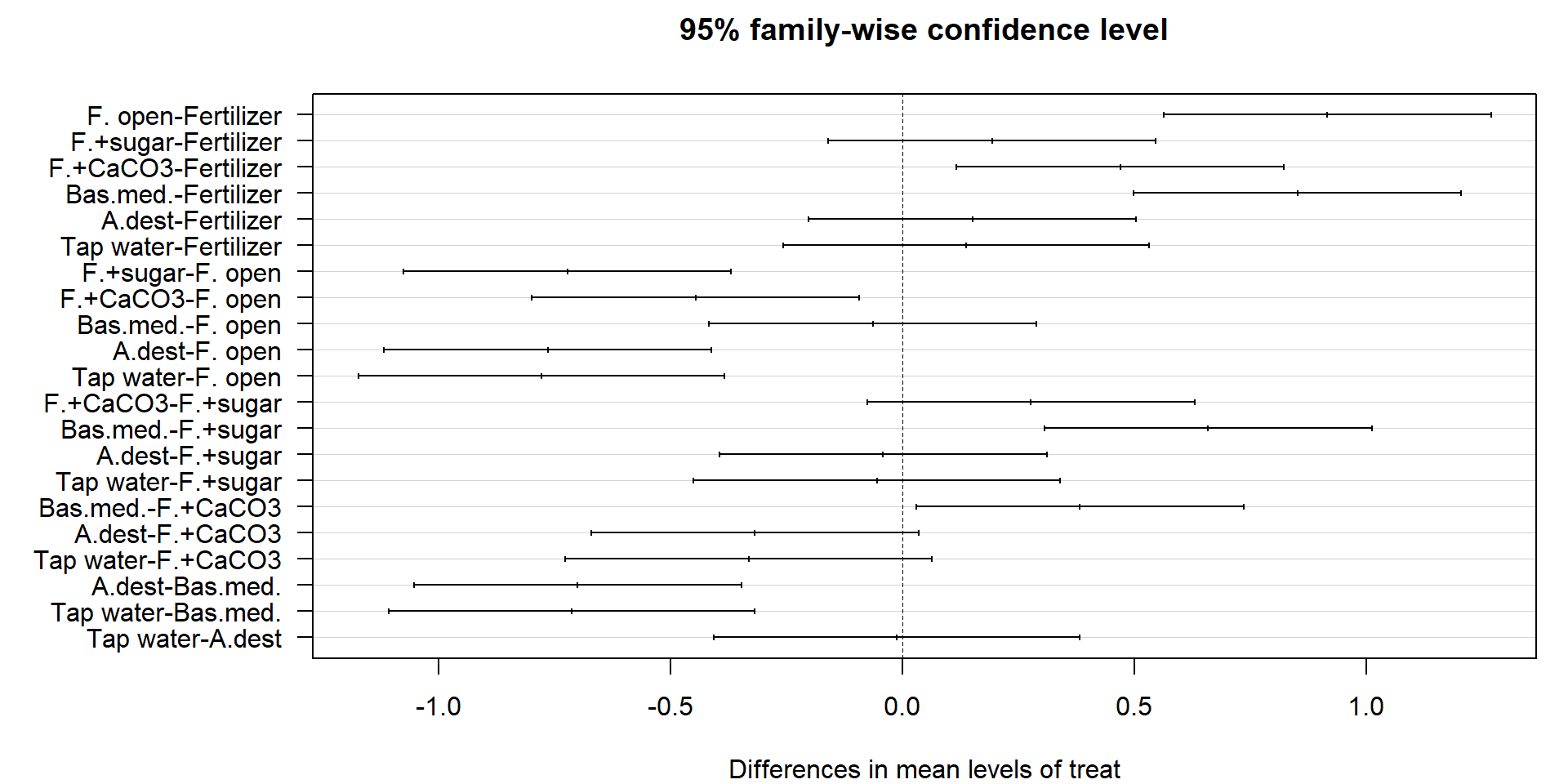
ANOVA Annahmen und Diagnostik
Für die ANOVA gelten dieselben Annahmen wie für das lineare Modell.
- Unabhängigkeit der Fehler
- Homogenität der Varianzen (Homoskedastizität)
- Annähernde Normalverteilung der Fehler
Grafische Diagnostik wird bevorzugt – sie ist robuster als numerische Tests.
Test der Varianzhomogenität
- Der F-Test ist nur für zwei Gruppen geeignet.
- Für mehr als zwei Gruppen: Bartlett-, Levene- oder Fligner-Killeen-Test.
- Empfohlen: Fligner-Killeen-Test - robust gegenüber Abweichung von Normalverteilung.
Test auf Normalverteilung
- Teste die Residuen, nicht die Rohdaten!
- Der Shapiro-Wilk-Test wird nicht mehr empfohlen:
- Bei kleinen Stichproben: hohe Wahrscheinlichkeit für Fehlertyp II
(keine Ablehnung, obwohl Unterschiede existieren). - Bei großen Stichproben: hohe Wahrscheinlichkeit für Fehlertyp I
(Ablehnung, obwohl Unterschiede vernachlässigbar sind.)
- Bei kleinen Stichproben: hohe Wahrscheinlichkeit für Fehlertyp II
- Empfohlen: Grafische Methoden – insbesondere der QQ-Plot.
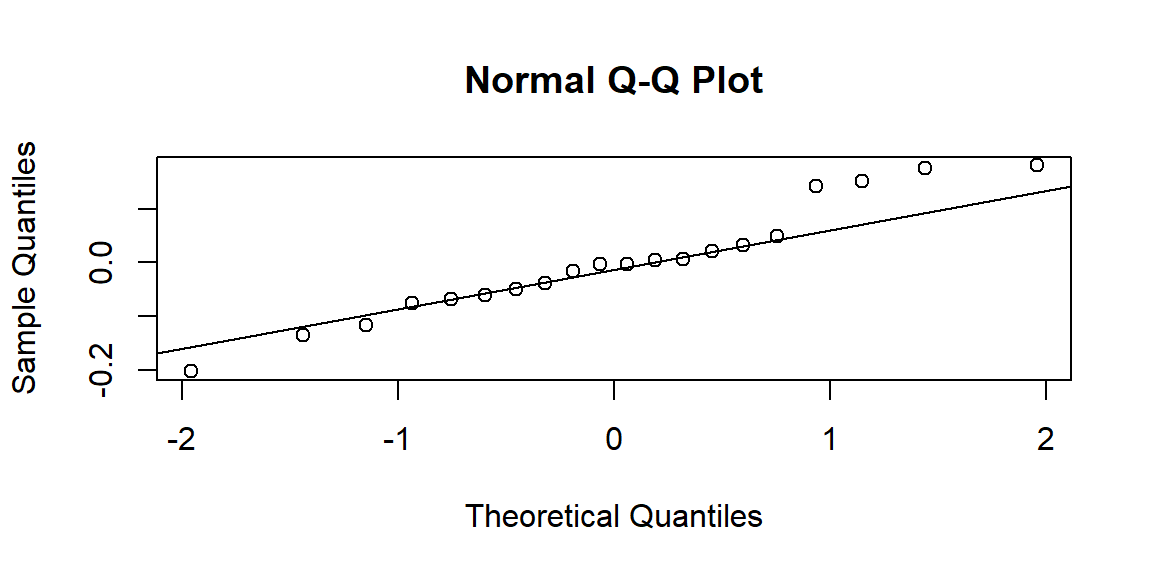
Moderne Sichtweise: Keine Vortests mehr!
Die traditionelle Praxis – Residuen zu testen, bevor man ANOVA durchführt –
ist nicht mehr zeitgemäß und führt zu gravierenden Problemen:
- Residuen sind erst nach Modellanpassung verfügbar.
- Bei Vortests müsste man sie „irgendwie vorher“ schätzen, oft durch Pooling.
- Häufig wurden Rohdaten statt Residuen getestet - Kardinalfehler.
- Die Methode stammt aus der Ära manueller Berechnungen.
Empfohlene Vorgehensweise:
- Prüfe, ob ANOVA grundsätzlich angemessen ist:
- Fragestellung,
- Unabhängigkeit,
- mögliche Nichtnormalität auf Grund des Versuchsdesigns
- Führe die ANOVA durch – ohne Vortests.
- Diagnostik im Anschluss – grafisch und kritisch (QQ-Plot, Residuals vs. Fitted).
Einfache ANOVA mit heterogenen Varianzen
- Erweiterung des Welch-Tests für \(\ge 2\) Stichproben
- in R
oneway.testgenannt.
One-way analysis of means (not assuming equal variances)
data: growth and treat
F = 115.09, num df = 6.0000, denom df = 4.6224, p-value = 6.57e-05- Der
oneway.testberücksichtigt ungleiche Varianzen durch gewichtete Mittelwerte und Freiheitsgrade und eine angepasste F-Statistik. - Die Varianzhomogenität ist wichtiger als die Normalverteilung – insbesondere bei unbalancierten Designs.
- Ein Varianzunterschied kann den F-Test stärker verzerren als leichte Abweichungen von der Normalverteilung.
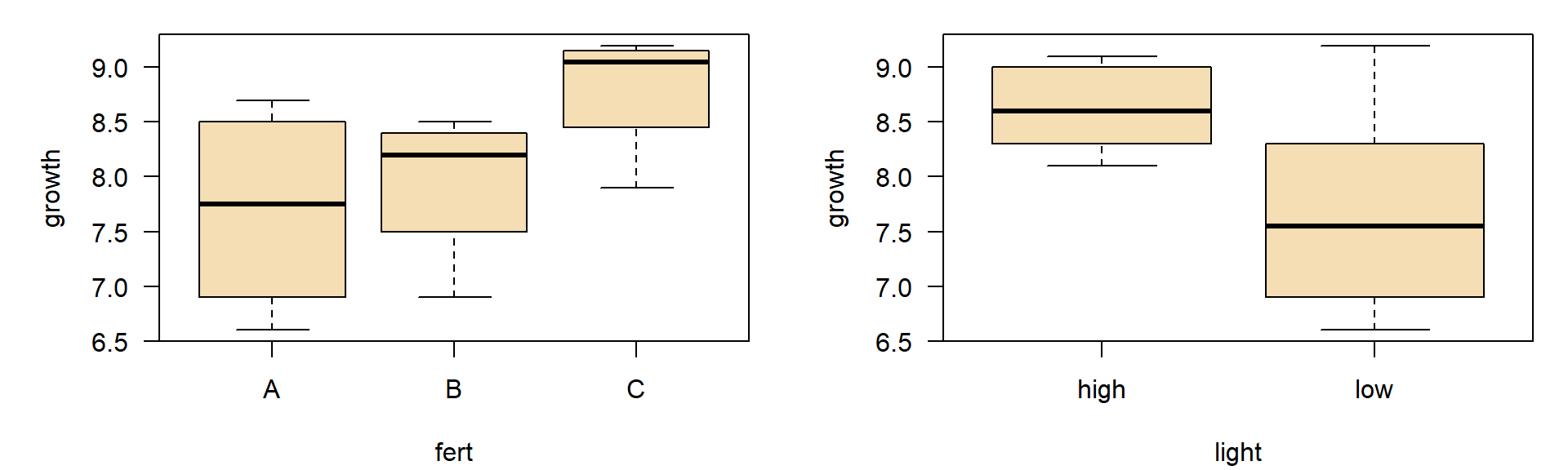
Zweifache ANOVA
- Beispiel aus einem Statistik-Lehrbuch (Crawley, 2002), angewandt auf einen neuen Kontext
- Auswirkungen von Dünger und Licht auf das Wachstum der Pflanzenhöhe in cm pro Zeit
| Dünger | helles Licht | schwaches Licht |
|---|---|---|
| A | 8.3 | 6.6 |
| A | 8.7 | 7.2 |
| B | 8.1 | 6.9 |
| B | 8.5 | 8.3 |
| C | 9.1 | 7.9 |
| C | 9.0 | 9.2 |
- faktorielles Experiment (mit Wiederholungen)
- für jede Faktorkombination mehr als eine Beobachtung
- Es können Wechselwirkungen identifiziert werden
Falls keine Wiederholungen: Keine Schätzung des Fehlers für Interaktionen - keine Identifizierung von Wechselwirkungen möglich.
Daten im Langformat eingeben
| No | growth | fert | light |
|---|---|---|---|
| 1 | 6.6 | A | low |
| 2 | 7.2 | A | low |
| 3 | 6.9 | B | low |
| 4 | 8.3 | B | low |
| 5 | 7.9 | C | low |
| 6 | 9.2 | C | low |
| 7 | 8.3 | A | high |
| 8 | 8.7 | A | high |
| 9 | 8.1 | B | high |
| 10 | 8.5 | B | high |
| 11 | 9.1 | C | high |
| 12 | 9.0 | C | high |
Beispiele für Modellformeln
| Modell Typ | Formel |
|---|---|
| Nullmodell | y ~ 1 |
| Einfache lineare Regression | y ~ x |
| Linear ohne Achsenschnittpunkt | y ~ x - 1 |
| Multiple Regression, keine Interaktion | y ~ x1 + x2 + x3 |
| Multiple Regression mit Interaktion | y ~ x1 * x2 * x3 |
| … ohne 3-fache Interaktion | y ~ x1 * x2 * x3 - x1 : x2 : x3 |
Transformiert mit I() (‘as is’) |
y ~ x + I(x^2) |
| Einfache ANOVA | y ~ f |
| ANOVA mit Interaktion | y ~ f1 * f2 |
| ANCOVA mit Interaktion | y ~ x * f |
| Verschachtelte ANOVA | y ~ x + (1 | a / b) |
GAM mit glättendem s |
y ~ s(x) + f |
y= Antwortvariable (abhängige, target)x= metrische Erklärungsvariable (Prädiktor, unabhängig)f= Faktorvariable (nominal)
Lineares Modell und ANOVA
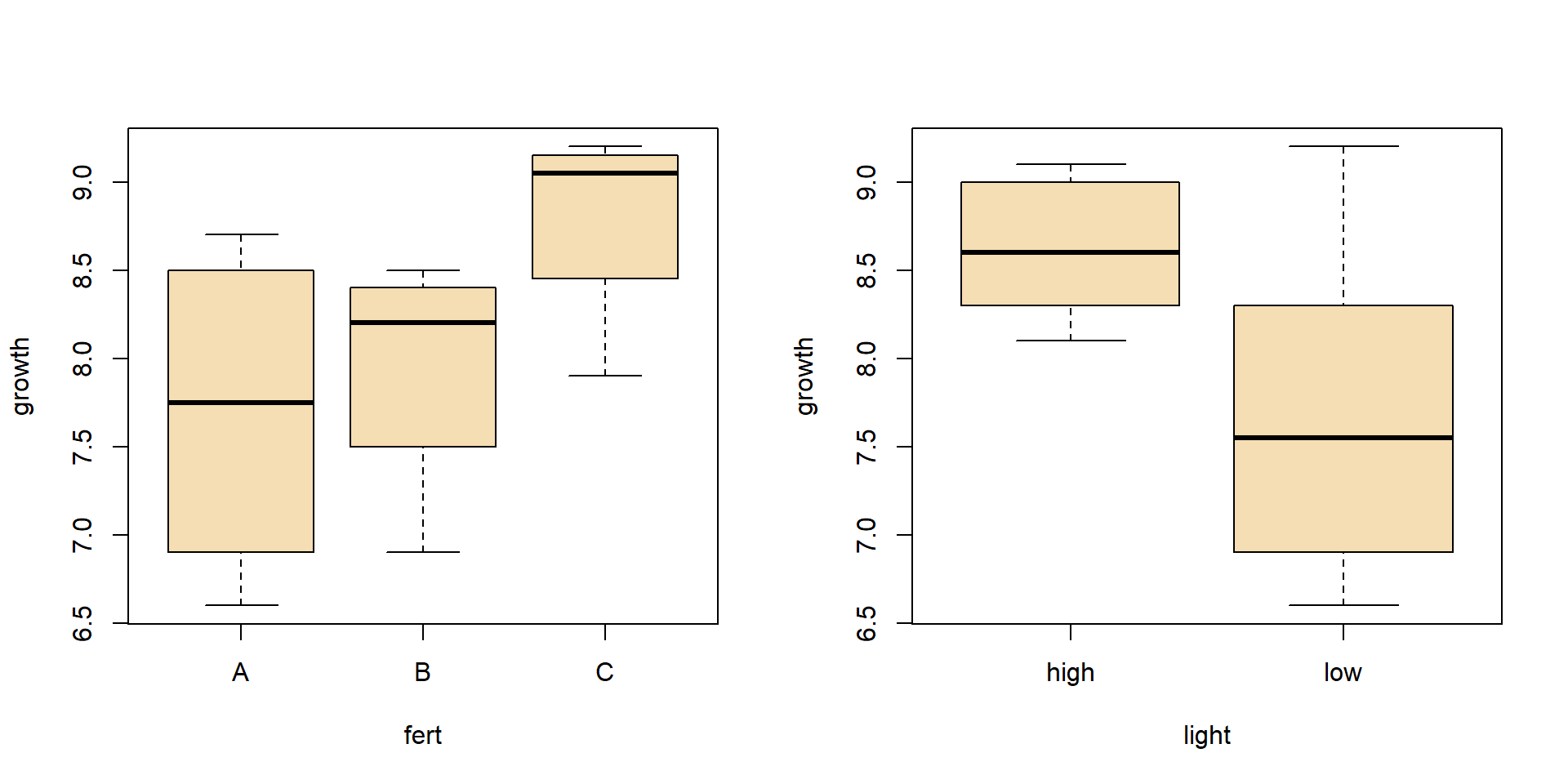
ANOVA
Analysis of Variance Table
Response: growth
Df Sum Sq Mean Sq F value Pr(>F)
light 1 2.61333 2.61333 7.2258 0.03614 *
fert 2 2.66000 1.33000 3.6774 0.09069 .
light:fert 2 0.68667 0.34333 0.9493 0.43833
Residuals 6 2.17000 0.36167
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Interaktionsplot
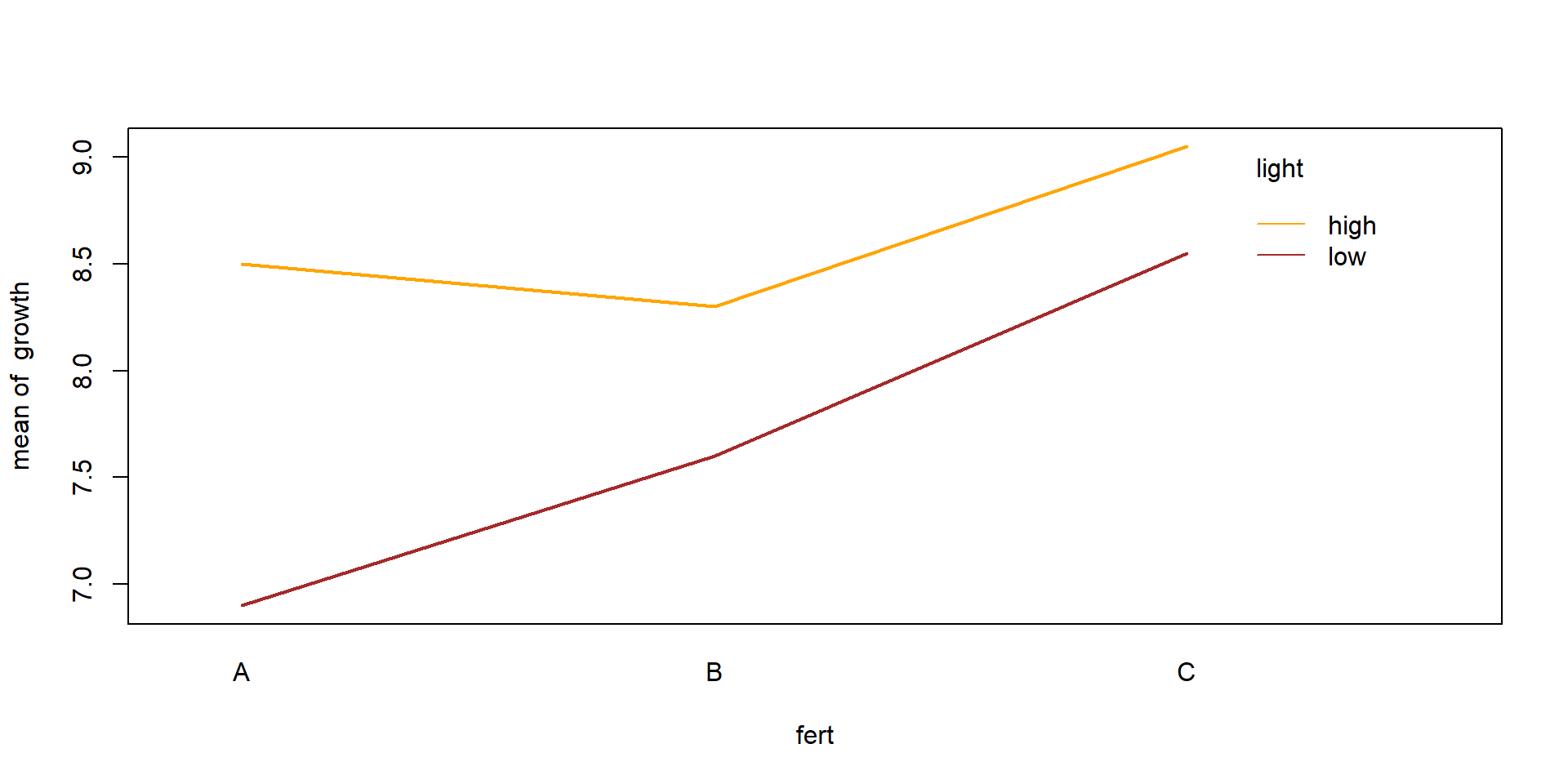
- Wenn die Linien nicht parallel sind, deutet es auf eine Wechselwirkung hin.
Diagnostik
- Unabhängigkeit der Messungen (innerhalb der Gruppen)
- Homogenität der Varianz der Residuen
- Normalverteilung der Residuen
Der Test der Annahmen benötigt Residuen des angepassten Modells.
\(\Rightarrow\) Passe zuerst das ANOVA-Modell an und prüfe dann, ob es richtig war!
Diagnostikinstrumente
- Boxplot (Gruppenvergleich)
- Residuen vs. Fitted
- Q-Q-Diagramm der Residuen
- Fligner-Killeen-Test (von manchen wird alternativ der Levene-Test empfohlen)
Diagnostik II
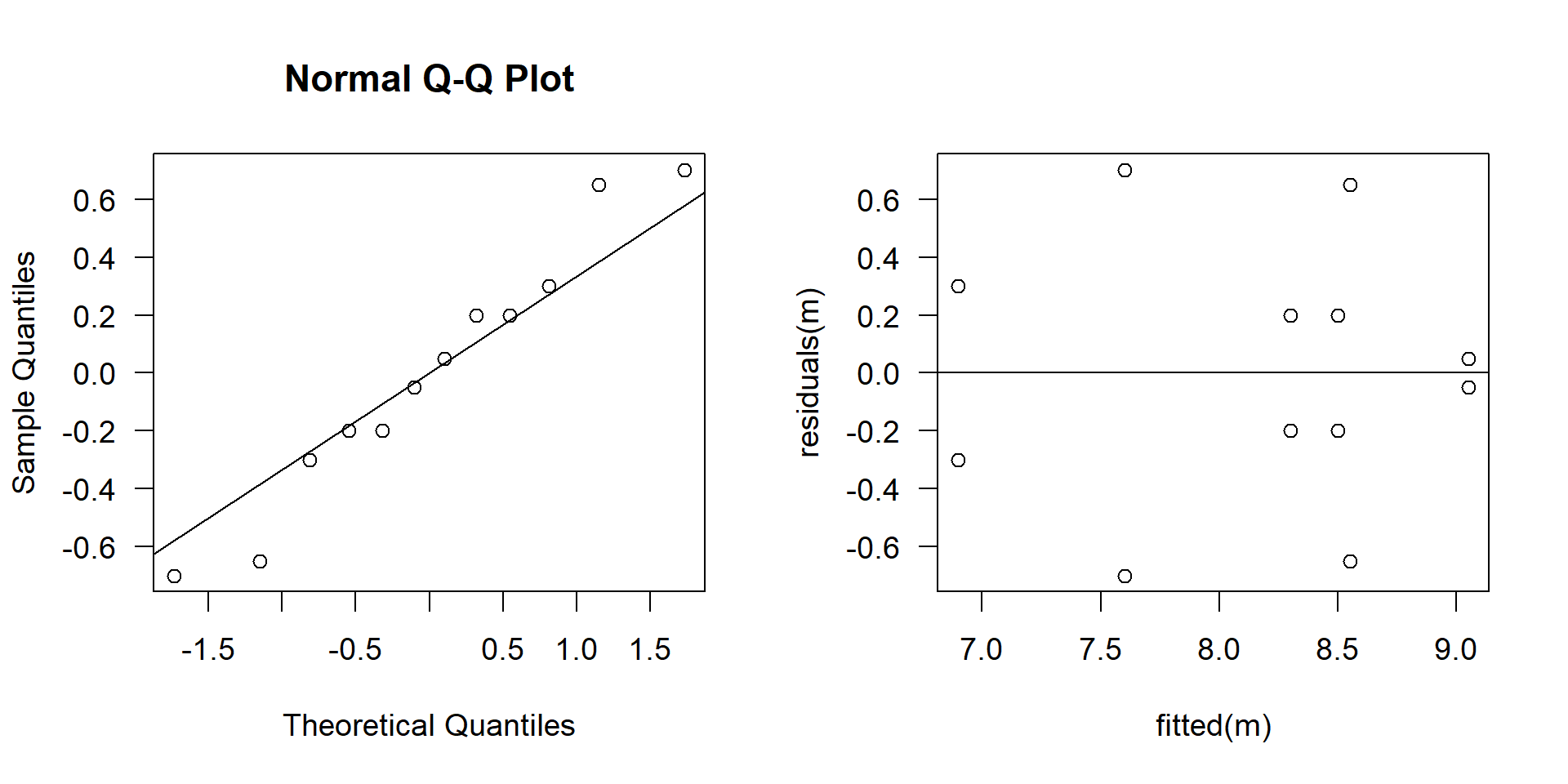
Fligner-Killeen test of homogeneity of variances
data: growth by interaction(light, fert)
Fligner-Killeen:med chi-squared = 10.788, df = 5, p-value = 0.05575Residuen: sehen in Ordnung aus und der p-Wert des Fligner-Tests ist noch ok.
Anmerkungen
Lineare Regression oder ANOVA?
- Im Wesentlichen dasselbe Modell - nur unterschiedliche Sichtweise
- Metrische unabhängige Variablen → lineares Modell
- Nominalskalierte unabhängige Variablen (Faktoren) → ANOVA
- Mischung aus beidem → ANCOVA
Pre-Tests sind theoretisch und praktisch fragwürdig
- Eine Nullhypothese \(H_0\) kann nicht bestätigt, sondern nur verworfen werden.
- Bei großen Stichproben ist die Normalität der Residuen nicht zwingend erforderlich.
- Wenn \(p\) in der Nähe des Schwellenwerts liegt und der Stichprobenumfang klein ist, bleiben wir im Ungewissen.
- All dies lässt sich nur durch sorgfältiges Nachdenken, Erfahrung und kritische Reflexion überwinden.
- Es ist immer eine gute Idee, die Ergebnisse mit anderen zu besprechen – nicht nicht um die Entscheidung zu delegieren, sondern um sie zu verstehen und zu verteidigen.
Sequentielles Holm-Bonferroni-Verfahren
- Auch bekannt als Holm-Korrektur (Holm, 1979)
- Einfach zu verwenden
- Kann auf jedes Problem mit Mehrfachtests angewendet werden
- Weniger konservativ als die normale Bonferroni-Korrektur, aber …
- … immer noch ein sehr konservativer Ansatz
- siehe auch Wikipedia
Algorithmus
- Wähle den kleinsten \(p\)-Wert aus allen \(n\) \(p\)-Werten
- Wenn \(p \cdot n < \alpha\) \(\Rightarrow\) signifikant, sonst
STOPP - Setze \(n - 1 \rightarrow n\), entferne das kleinste \(p\) aus der Liste und gehe zu Schritt 1.
Der Algorithmus ist sequenziell
- Er prüft die Hypothesen in der Reihenfolge der kleinsten \(p\)-Werte.
Beispiel
Wachstumsrate pro Tag (\(d^{-1}\)) von Blaualgenkulturen (Pseudanabaena) nach Zugabe toxischer Peptide einer anderen Blaualge (Microcystis).
Die ursprüngliche Hypothese:
- Microcystin LR (MCYST) oder ein Derivat davon (Substanz A) hemmt das Wachstum.
Ansatz 1: einfache ANOVA
Analysis of Variance Table
Response: mu
Df Sum Sq Mean Sq F value Pr(>F)
treat 2 0.00053293 2.6647e-04 8.775 0.004485 **
Residuals 12 0.00036440 3.0367e-05
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Ansatz 2: multiple t-Tests mit sequentieller Bonferroni-Korrektur
Wir trennen den Datensatz in einzelne Teilmengen:
und führen 3 t-Tests durch:
p-Werte ohne Korrektur:
Mit Holm-Korrektur:
Schlussfolgerungen
Statistische Methoden
- Beim Holm-korrigierten t-Test bleibt nur ein einziger p-Wert (MCYST vs. Subst A) signifikant, bei der ANOVA sind es zwei.
- ANOVA berücksichtigt Struktur der Daten und die Abhängigkeit der Vergleiche.
- Holm-Methode konservativer als TukeyHSD \(\Rightarrow\) ANOVA + Posthoc ist zu bevorzugen.
Interpretation hinsichtlich der ursprünglichen Hypothese
MCYST und Subst A hemmen das Wachstum von Pseudanabaena nicht.
Im Gegenteil: Subst A stimuliert das Wachstum.
Widerspruch zur Erwartung: der biologische Grund wurde 10 Jahre später entdeckt.
Wichtig: Statistische Signifikanz sagt nichts über die biologische Relevanz aus.
Falsche Hypothesen können zu falschen Erwartungen führen - oder zu neuen Entdeckungen.
Mehr dazu in Jähnichen et al. (2001), Jähnichen et al. (2007), Jähnichen et al. (2011), Zilliges et al. (2011), Dziallas & Grossart (2011), Wei et al. (2024)
ANCOVA
Statistische Frage
- Vergleich von Regressionslinien zwischen Gruppen
- Ähnlich wie bei der ANOVA, enthält aber auch metrische Variablen (Kovariaten)
- Haben Gruppen unterschiedliche Anfangswerte (Intercept) und/oder unterschiedliche Steigungen (Slope)?
Beispiel
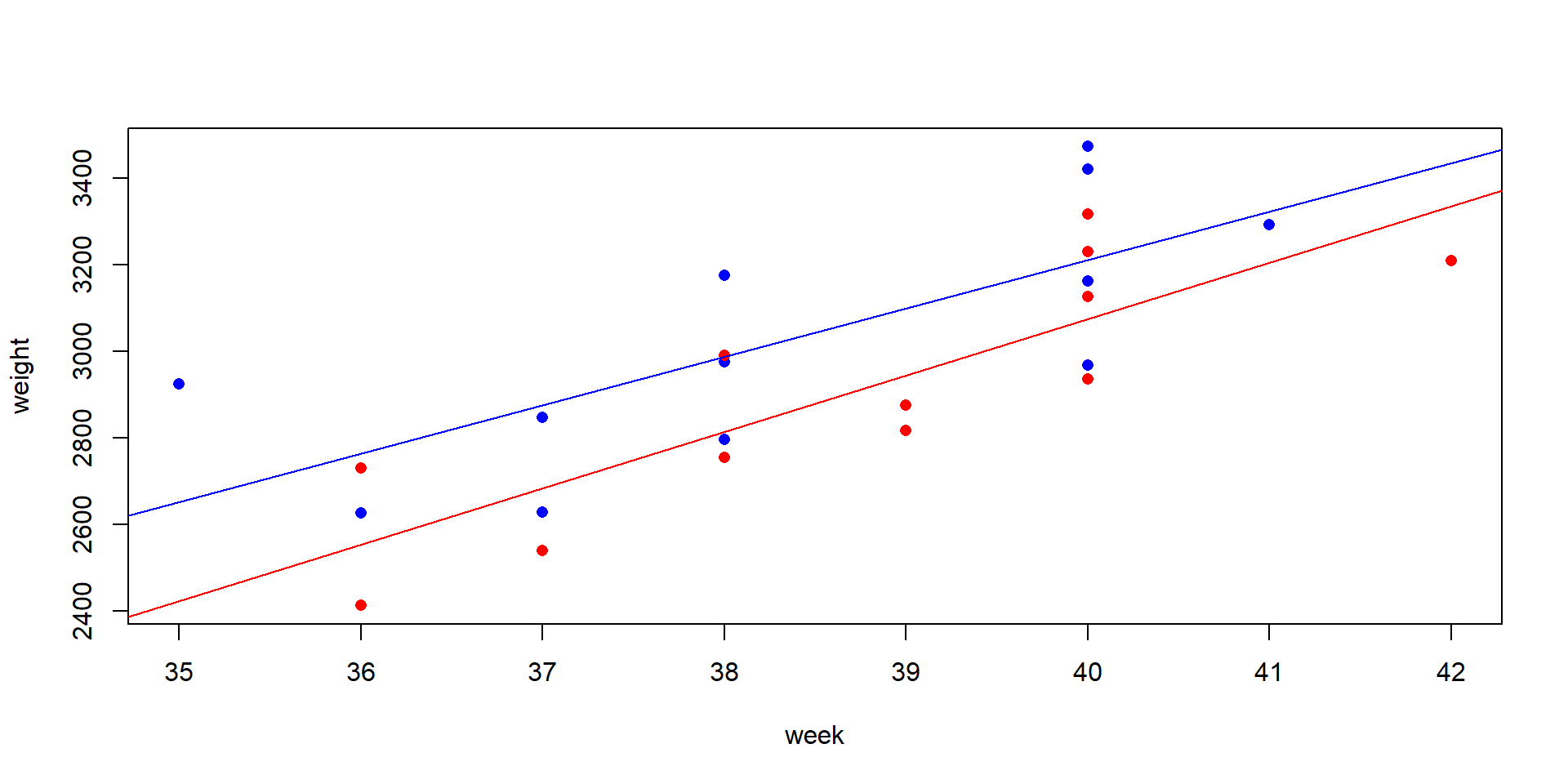
Annette Dobsons Geburtsgewichts-Daten – aus einem Statistik-Lehrbuch (Dobson, 2013): Geburtsgewicht von Jungen und Mädchen in Abhängigkeit von der Schwangerschaftswoche.
Der Datensatz zum Geburtsgewicht
Dieser Datensatz ist künstlich – nicht realistisch, aber ideal für die Demonstration.
Er ist in der R-Demo demo(lm.glm) enthalten.
## Daten zum Geburtsgewicht siehe stats/demo/lm.glm.R
dobson <- data.frame(
week = c(40, 38, 40, 35, 36, 37, 41, 40, 37, 38, 40, 38,
40, 36, 40, 38, 42, 39, 40, 37, 36, 38, 39, 40),
weight = c(2968, 2795, 3163, 2925, 2625, 2847, 3292, 3473, 2628, 3176,
3421, 2975, 3317, 2729, 2935, 2754, 3210, 2817, 3126, 2539,
2412, 2991, 2875, 3231),
gender = gl(2, 12, labels = c("M", "F")) # generate factor levels
)Warum nicht einfach einen t-Test durchführen?
Two Sample t-test
data: weight by gender
t = 0.97747, df = 22, p-value = 0.339
alternative hypothesis: true difference in means between group M and group F is not equal to 0
95 percent confidence interval:
-126.3753 351.7086
sample estimates:
mean in group M mean in group F
3024.000 2911.333 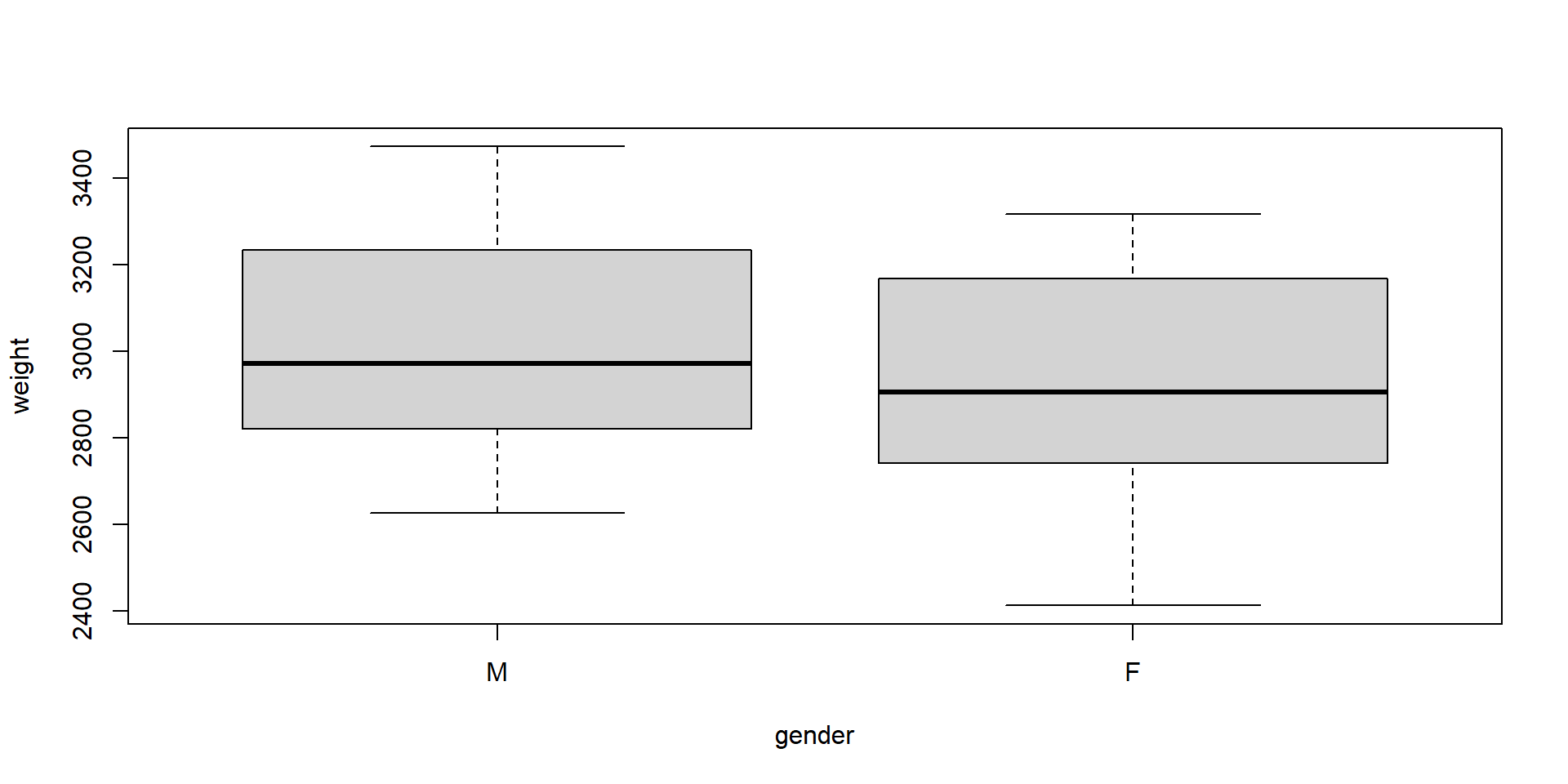
- Der t-Test zeigt keine signifikante Differenz und der Boxplot zeigt starke Überlappung.
- Aber: Der t-Test ignoriert die Schwangerschaftswoche, eine wichtige Kovariate.
ANCOVA: Berücksichtigung der Kovariate
Analysis of Variance Table
Response: weight
Df Sum Sq Mean Sq F value Pr(>F)
week 1 1013799 1013799 31.0779 1.862e-05 ***
gender 1 157304 157304 4.8221 0.04006 *
week:gender 1 6346 6346 0.1945 0.66389
Residuals 20 652425 32621
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1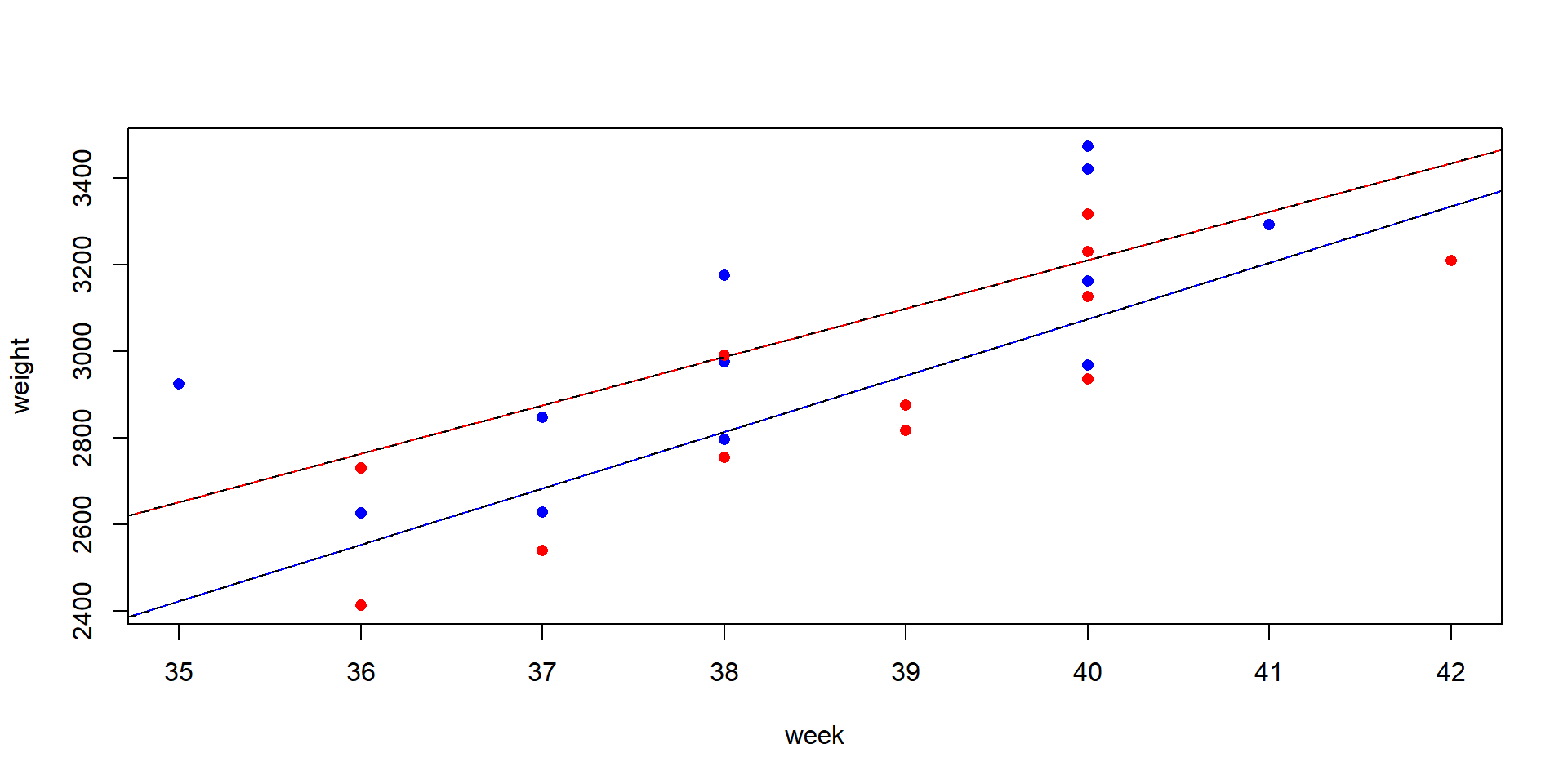
Beispiel: Aus Gründen, die wir später besprechen, muss die Kovariate week als erstes in der Modellformel stehen.
Tücken der ANOVA/ANCOVA
Bisher beschriebene Tücken von ANOVA und ANCOVA
- Heterogenität der Varianz
- p-Werte können verzerrt sein (d.h. irreführend oder falsch)
- Verwendung einer einfachen ANOVA für ungleiche Varianzen (in R:
oneway.test)
- Unbalanzierter Fall
- Ungleiche Anzahl von Stichproben für jede Faktorkombination
- \(\rightarrow\) ANOVA-Ergebnisse sind von der Reihenfolge der Faktoren in der Modellformel abhängig.
- Klassische Lösung: Typ II oder Typ III ANOVA
- Moderner Ansatz: Modellselektion und Likelihood-Ratio-Tests
Typ II und Typ III ANOVA
- Funktion
Anova(mit GroßbuchstabeA) im Paket car - Hilfeseite der Funktion
Anova:
Type-II tests are calculated according to the principle of marginality, testing each term after all others, except ignoring the term’s higher-order relatives; so-called type-III tests violate marginality, testing each term in the model after all of the others.
- Schlussfolgerung: Verwende Typ II und nicht Typ III.
- Interpretiere keine einzelnen Haupteffekte, wenn eine signifikante Wechselwirkung vorliegt.
Typ II ANOVA: Beispiel
Anova Table (Type II tests)
Response: growth
Sum Sq Df F value Pr(>F)
light 2.61333 1 7.2258 0.03614 *
fert 2.66000 2 3.6774 0.09069 .
light:fert 0.68667 2 0.9493 0.43833
Residuals 2.17000 6
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Modellselektion -
ein Paradigmenwechsel
Modellkandidaten und Auswahl eines optimalen Modells
Problem:
- Bei komplexen ANOVA-Modellen hängen die p-Werte von der Anzahl (und manchmal von der Reihenfolge) der einbezogenen Faktoren und Wechselwirkungen ab.
- Der H0-basierte Ansatz wird eventuell verwirrend, z.B. wegen widersprüchlicher p-Werte.
Alternativer Ansatz:
- Modellselektion – nutzt das Prinzip der Parsimonie
Es werden verschiedene Modellkandidaten verglichen:
- Modell mit allen potentiellen Effekten → vollständiges Modell
- Weglassen einzelner Faktoren → reduzierte Modelle (mehrere!)
- Keine Einflussfaktoren (nur Mittelwert) → Nullmodell
- Welches Modell ist das beste? → minimales adäquates Modell
Wie können wir messen, welches Modell das beste ist?
Kompromiss zwischen Modellanpassung und Modellkomplexität (Anzahl der Parameter, k).
- Güte der Anpassung: Likelihood L – wie gut die Daten zu einem bestimmten Modell passen.
- Log Likelihood: macht das Kriterium additiv.
- AIC (Akaike Information Criterion):
\[AIC = −2 \ln(L) + 2k\]
- BIC ( Bayesian Information Criterion), berücksichtigt den Stichprobenumfang (\(n\)):
\[BIC = −2 \ln(L) + k \cdot \ln(n)\]
Das Modell mit dem kleinsten AIC (oder BIC) ist das minimal adäquate Modell.
Modellselektion und Likelihood-Ratio-Tests
Ansatz
- Mehrere Modelle einzeln anpassen
- Modelle paarweise mit ANOVA (Likelihood Ratio Test) vergleichen
Daten und Beispiel
m3 <- lm(growth ~ fert * light, data=plants) # f1 + f2 + f1:f2
m2 <- lm(growth ~ fert + light, data=plants) # f1 + f2
anova(m3, m2)Analysis of Variance Table
Model 1: growth ~ fert * light
Model 2: growth ~ fert + light
Res.Df RSS Df Sum of Sq F Pr(>F)
1 6 2.1700
2 8 2.8567 -2 -0.68667 0.9493 0.4383- Likelihood-Ratio-Test vergleicht zwei Modelle (
anovain R mit > 1 Modell) - Modell mit Interaktion (
m3) nicht signifikant besser als Modell ohne Interaktion (m2).
AIC-basierte Modellselektion
- Der paarweise Modellvergleich ist umständlich, insbesondere bei einer großen Anzahl von Modellen.
- Lösung: Erstelle eine Menge von Kandidatenmodellen
- Verwende kleinstes AIC, um das minimal angemessene Modell auszuwählen.
m3 <- lm(growth ~ light * fert, data = plants) # Gesamtmodell
m2 <- lm(growth ~ light + fert, data = plants)
m1a <- lm(growth ~ fert, data = plants)
m1b <- lm(growth ~ light, data = plants)
m0 <- lm(growth ~ 1, data = plants) # Nullmodell
AIC(m0, m1a, m1b, m2, m3) df AIC
m0 2 33.38238
m1a 4 32.62699
m1b 3 30.72893
m2 5 26.83151
m3 7 27.53237Anmerkungen
- AIC-Werte sind bis zu einer additiven Konstante definiert
- \(\rightarrow\) Absolute Werte können je nach Software oder Methode variieren.
- \(\Rightarrow\) Betrachte die Differenzen, nicht die absoluten Werte.
- Faustregel: die „AIC-Einheit“ ist 2, Unterschiede \(\approx\) 2.0 \(\rightarrow\) geringe Bedeutung
Schrittweise Modellselektion (automatisch)
Automatische Modellselektion
Start: AIC=-8.52
growth ~ fert * light
Df Sum of Sq RSS AIC
- fert:light 2 0.68667 2.8567 -9.2230
<none> 2.1700 -8.5222
Step: AIC=-9.22
growth ~ fert + light
Df Sum of Sq RSS AIC
<none> 2.8567 -9.2230
- fert 2 2.6600 5.5167 -5.3256
- light 1 2.6133 5.4700 -3.4275Modell mit dem kleinsten AIC \(\rightarrow\) optimales Modell.
Zum Vergleich: klassische ANOVA
Ergebnisse des Beispiels:
- optimales Modell (
opt), enthält beide Faktoren (fert,light), aber keine Interaktion. - Die Modellselektion identifiziert
fertundlightals wichtige Erklärungsvariablen. - im Gegensatz zur klassischen ANOVA-Tabelle, in der nur
lightsignifikant war, zeigt die Modellselektion, dass auchfertzur Erklärung beiträgt.
Signifikanztests vs. Modellselektion
- Das Konzept der Modellselektion ersetzt die p-Wert-basierte Statistik, zumindest teilweise.
- Einige Autoren raten davon ab, p-Werte in diesem Kontext noch zu verwenden.
- Andere empfehlen einen Kompromiss: p-Werte als Hilfsmittel, nicht als Entscheidungskriterium.
Derzeitige Empfehlung
Wenn p-Werte benötigt werden, vergleiche das optimale Modell mit reduzierten Modellen:
- Interpretiere p-Werte mit Vorsicht:
- Sie sind abhängig von der Modellstruktur.
- Sie können irreführend sein, wenn das Modell nicht optimal ist.
- Gib auch Effektstärken und Vertrauensintervalle an!
Zusammenfassung des Kapitels ANOVA
- Lineare Modelle bilden die Grundlage für viele statistische Methoden
- Lineare Regression
- ANOVA/ANCOVA anstelle von Mehrfachtests
- MANOVA, GLM, GAM, GLMM, . . .
- ANOVA ist leistungsfähiger als Mehrfachtests:
- vermeidet \(\alpha\)-Fehlerinflation
- identifiziert Interaktionseffekte
- eliminiert Kovariate
- ein großes Experiment benötigt weniger Stichprobengröße als viele kleine Experimente
- Modellselektion vs. p-Wert-basierte Tests
- Paradigmenwechsel: AIC anstelle von p-Werten
- zuverlässiger, besonders bei unausgewogenen oder komplexen Designs
- erweiterbar auf GLM, GAM, LME, GLMM, …
- aber: p-Wert-basierte Tests sind manchmal leichter zu verstehen
Selbststudium: Lies das Paper von Johnson & Omland (2004) zum Thema Modellselektion.
Vermeide Manipulation von p-Werten
Experimente NICHT wiederholen, bis ein signifikanter p-Wert gefunden wird!
Leek et al. (2017) in Nature:
“… Nature asked influential statisticians to recommend one change to improve science. The common theme? The problem is not our maths, but ourselves.”:
Five ways to fix statistics. Comment on Nature
- Jeff Leek: Adjust for human cognition
- Blakeley B. McShane & Andrew Gelman: Abandon statistical significance
- David Colquhoun: State false-positive risk, too
- Michèle B. Nuijten: Share analysis plans and results
- Steven N. Goodman: Change norms from within
The “ASA Statement …”
Das ASA Statement on Statistical Significance and P-Values der American Statistical Association (Wasserstein & Lazar, 2016) befasst sich mit der Bedeutung des p-Werts, ohne ihn komplett in Frage zu stellen. Es definiert 6 Prinzipien:
- P-values can indicate how incompatible the data are with a specified statistical model.
- P-values do not measure the probability that the studied hypothesis is true, or the probability that the data were produced by random chance alone.
- Scientific conclusions and business or policy decisions should not be based only on whether a p-value passes a specific threshold.
- Proper inference requires full reporting and transparency.
- A p-value, or statistical significance, does not measure the size of an effect or the importance of a result.
- By itself, a p-value does not provide a good measure of evidence regarding a model or hypothesis.
Die Details sind im Statement weiter ausgeführt.
Selbststudium: Lies dazu neben Wasserstein & Lazar (2016) auch Hurlbert et al. (2019).